構築一個重組蛋白表現質體 (recombinant protein expressing clone), 主要包含了下列幾個步驟:
(1) 設計適當的引子(forward & reverse primers).
(2) 利用PCR儀器促使DNA template與我們設計的引子形成配對, 再藉由熱穩定性高的DNA polymerase將引子延長, 進而放大含有目標蛋白基因序列之DNA片段數量.
(3) 取得含有目標基因的DNA片段之後, 加入限制酵素 (restriction enzyme) 對DNA片段進行切割.
(4) 加入同樣經過限制酶切割的表現載體 (e.g. pET, pQE vectors…) 與前述DNA片段, 以DNA 黏合酶 (ligase) 進行互補切位的黏合反應.
(5) 將黏合反應後的產物transform至host cell中複製.
(6) 抽取重組質體進行定序確認 (sequencing).
在進行cloning流程之前最好先確認下列事項:
1. 確認具有目標蛋白完整的DNA (gene)序列與DNA template.
2. 利用簡易的生物資訊網站
(e.g. ExPASy, Biology workbench…等) 將目標蛋白胺基酸序列進行初步分析.根據所獲得的資訊,
評估是否要將目標蛋白整段 gene都放入表現質體當中. 例如目標蛋白具有穿膜區域(transmembrane
helices)或訊息胜肽(N-terminal signal peptide)…等. 這些因素都會影響重組蛋白質的溶解度與接下來的實驗設計,
宜多加留意.
3. 利用目標蛋白胺基酸序列進行二級結構預測,倘若其N端或C端區域若有長片段unstructured區域, 在不影響重組蛋白質活性之原則下,建議將其去除, 這會有利於接下來的蛋白質晶體培養.
4. 根據實驗的目的,
挑選適當的表現載體 (參考實驗室 vector & host list), 並確認其MCS (multiple cloning
site) 區域中具有至少兩個restriction enzyme cutting-site (RE site) 不曾在目標蛋白的DNA
sequence中出現.
(1) 設計引子(primer)
有上述的初步分析, 我們就可以開始著手設計 forward 和 reverse 引子.
一條引子除了5’端的RE-site sequence之外, 還包括了3’ 端會與DNA template產生base-pairing 的序列
(如下圖).

Duplex DNA template
RE
site的選用, 取決於各種載體MCS (multiple cloning sites) 區域中所提供的cutting-sites,
儘可能挑選常見, 切割效率好的RE種類. 至於3’端Base-pairing 序列長短大小則會影響primer的專一性. 一般來說18-25
個nucleotide長度的互補區域搭配以G 或 C作為結尾的設計, 即可提供引子良好的專一性. 設計完primer之後就可以向廠商訂購,
幫忙合成我們所需要的引子.
(2) PCR 反應
PCR 反應所需要添加的反應物及其濃度大致如下列:
1. DNATemplate (plasmid DNA 10 pg~100 ng/ genomic DNA 50 ng~100 ng )
2.Forward Primer 0.2~0.5 mM
Reverse Primer 0.2~0.5 mM
3. each dNTP
0.2 mM
4.10x buffer
5ul
5.pfu DNA polymerase
1ul
6.add dH2O
to
50 ul
 Total reaction volume 50 ul
Total reaction volume 50 ul
將反應物放置於PCR tube, 開始進行PCR的基本反應步驟:
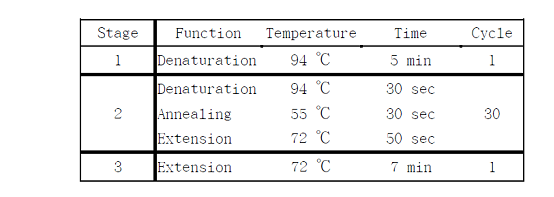
以總體積 50 ml 的 PCR 反應為例, 藉由 PCR machine 執行 25~28 個
melting-annealing-extension 的溫控循環 (cycle)之後, DNA polymerase 所催化的聚合反應,
就能生產足夠的 DNA片段產物.
(3) 限制酶切割反應 (restriction enzyme digestion)
1. 將限制酶切割所需要的反應物依序加入1.5 ml 小離心管中 (以總反應體積20 ml為例)
DNA (PCR product or vector/plasmid) 2~10 mg
10x restriction buffer
2 ul
Restriction-enzyme
< 2 ul (no more than 10% of final volume)
Add dH2O
to 20 ul
Total reaction volume
20 ul
2. 參考各種限制酶最適合的反應溫度進行作用, 例如: 37℃水浴作用 1~數個小時.
3. 以 PCR clean-up kit 回收 DNA.
(4) DNA黏合反應 (DNA ligation)
將切割過的DNA片段 (insert) 與經過同樣限制酶處理的載體(Vector) 以不同莫耳數比例 (1:1, 3:1,或6:1) 進行黏合反應,37℃ 1hr 或 16℃ overnight. (以總反應體積20 ml為例)
DNA insert/Vector molar ratio 1, 3, or 6
10x ligation buffer
2ul
DNA ligase
1ul
Add dH2O
to 20ul
Total reaction volume 20ul
註: insert+ vector 的DNA數量不要超過 300 ng
(5) Transformatio

(6) 抽取質體送定序
從前述plate上挑取單一菌落, 加入50ml LB與適當抗生素
進行37 ℃ overnight培養, 接著抽取質體.


Bookmark/Search this post with:



 Total reaction volume 50 ul
Total reaction volume 50 ul